近日,由云南大学孙艳波研究员和中国科学院昆明动物研究所吴东东研究员领衔的科研团队,圆满完成经典模式生物黑腹果蝇Canton S品系的近完整端粒到端粒基因组(Dm.nT2T)构建工作。该项研究成果以《Near Complete Assembly of Drosophila melanogaster Canton S strain genome》为题,在《Nature Communications》期刊上正式发表(https://doi.org/10.1038/s41467-025-67031-w)。云南大学刘彦男博士(现就职于中国科学院昆明动物研究所)和高建军副研究员为并列第一作者,孙艳波研究员和吴东东研究员为并列通讯作者。此项工作得到了云南省基础研究计划重大项目,云南大学“双一流”建设经费、及国家重点研发(子课题)的支持。该成果标志着对果蝇这一核心模型生物的基因组解析达到了前所未有的完整度与准确度,是基因组学研究领域的一项重要突破。
黑腹果蝇(Drosophila melanogaster)是遗传学、发育生物学和神经科学研究的基石。然而,其现行参考基因组(ISO-1品系,Release 6)受限于早期测序技术,在高度重复的着丝粒、端粒等序列重复区仍存在大量缺口(gap),制约了对其基因组结构完整性、进化动态及非编码调控元件的深入探索。为攻克这一难题,研究团队采用了多平台测序数据整合策略:PacBio HiFi测序:提供高精度(平均Q28.9)长读长序列(~7.1 kb),用于精准校正。Oxford Nanopore超长读长测序:产生平均长度达26.5 kb的序列,跨越大型重复单元。Hi-C染色质构象捕获技术:提供染色体三维互作信息,辅助完成染色体水平组装。
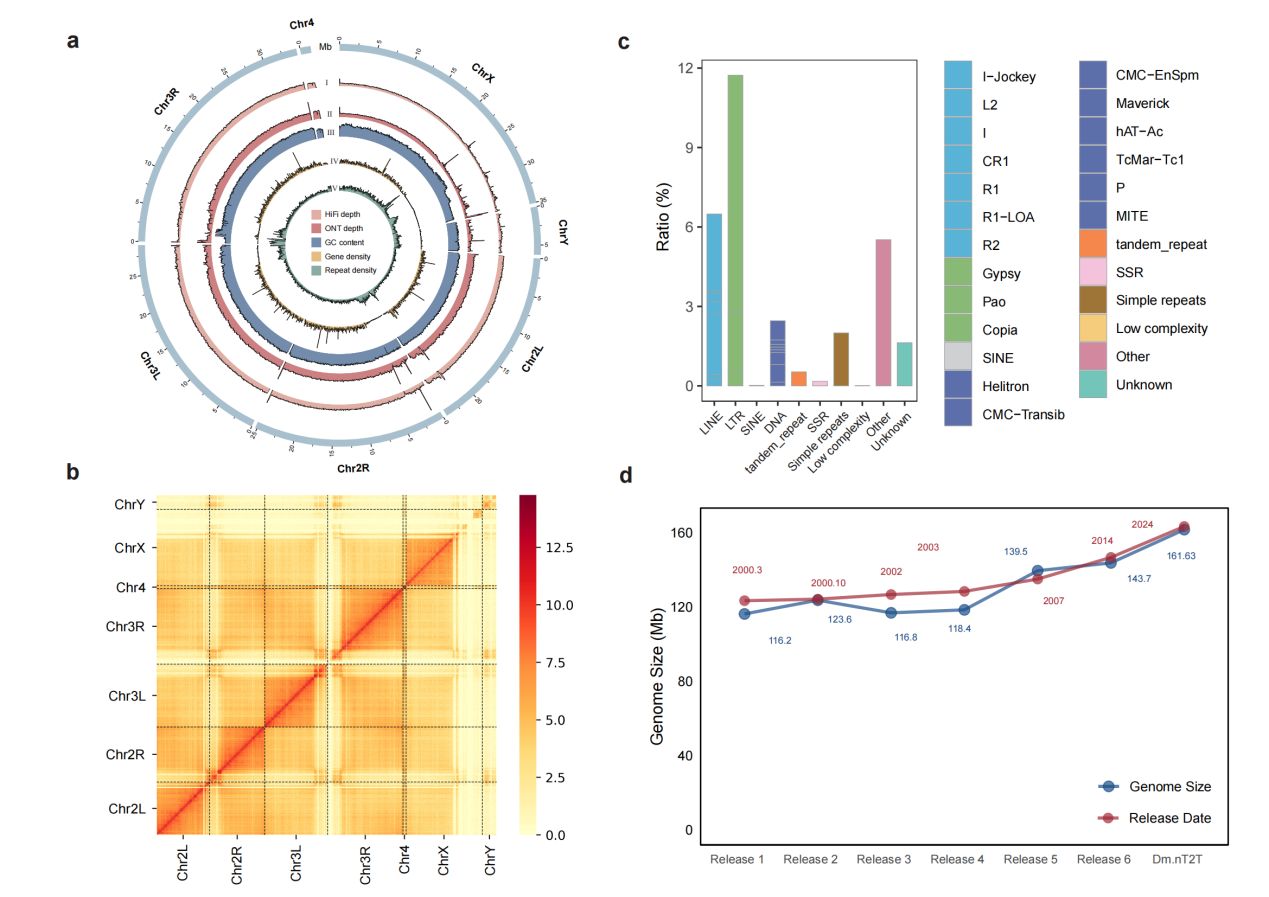
图1.Dm.nT2T基因组组装特征环形图,展示测序深度、GC含量、基因与重复序列密度分布
通过集成上述数据,并使用NextDenovo、NextPolish等流程进行组装,团队最终获得了命名为Dm.nT2T的基因组组装(图1)。该组装总长达161.63 Mb,包含7条染色体,Contig N50达到21.93 Mb(表1)。该组装在连续性和完整性上显著提升,经Mercury评估的组装质量值(QV)达40.24,首次将果蝇的rDNA簇与组蛋白基因簇等复杂重复区域完整、准确定位到染色体特定位置。

通过与以往参考基因组版本的比较分析后得到以下突破性发现:(1)首次在果蝇中鉴定出SINE转座子:重复序列分析显示,Dm.nT2T中SINE(短散在核元件)占比0.02%。这是首次在黑腹果蝇基因组中明确报告此类元件。序列分析表明,它们与已知的DINE-1元件相似度极低(16-30%),可能代表了果蝇中一个独立进化谱系的SINE家族。(2)系统解析结构变异(SV):通过与ISO-1 R6参考基因组的比较,研究团队在Canton S品系中鉴定出7,989个结构变异,包括2,862个插入、3,208个缺失及大量倒位、易位等。这些SV显著富集于端粒、着丝粒区域以及Y染色体,其中79.57%的插入/缺失(INDEL)长度超过100 bp(图2),远超出短读长测序的检测能力,凸显了高质量参考基因组对发现大型变异的重要性。
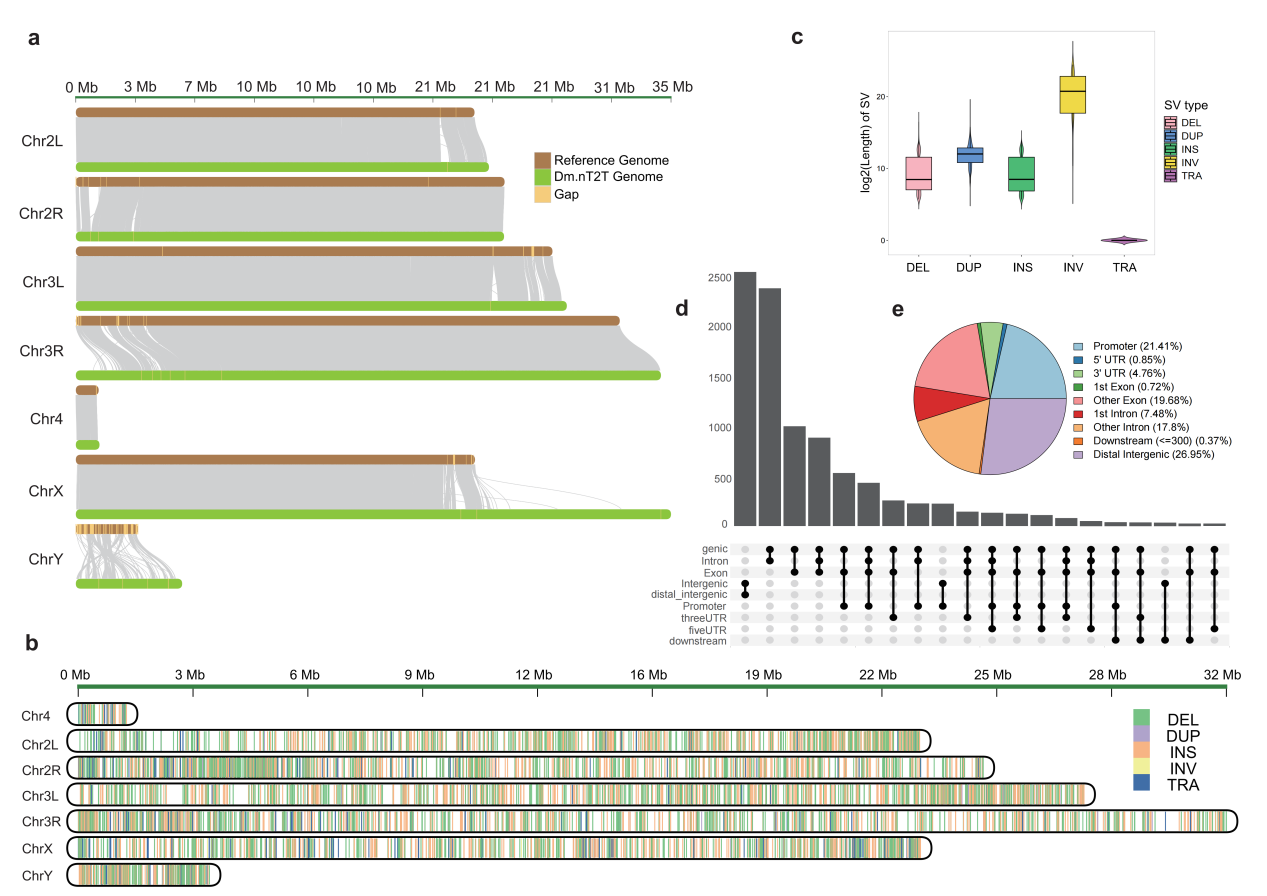
图2.Dm.nT2T与R6基因组共线性比对及结构变异染色体分布图谱
(3)精准定位端粒与着丝粒:(1)果蝇端粒由HeT-A、TART、TAHRE等逆转录转座子维持。Dm.nT2T中鉴定出6个端粒区域,总长度达297 kb,远超R6中的74.3 kb(图3)。其中X染色体左臂端粒最长,为76.24 kb。(2)着丝粒:结合串联重复查找(TRF)和CENP-A组蛋白的ChIP-seq数据,团队精确界定了各染色体的着丝粒区间(图3)。这些区域富含5-12 bp的短卫星重复序列,其中5 bp重复单元拷贝数高达29万次,构成了果蝇着丝粒的核心特征。
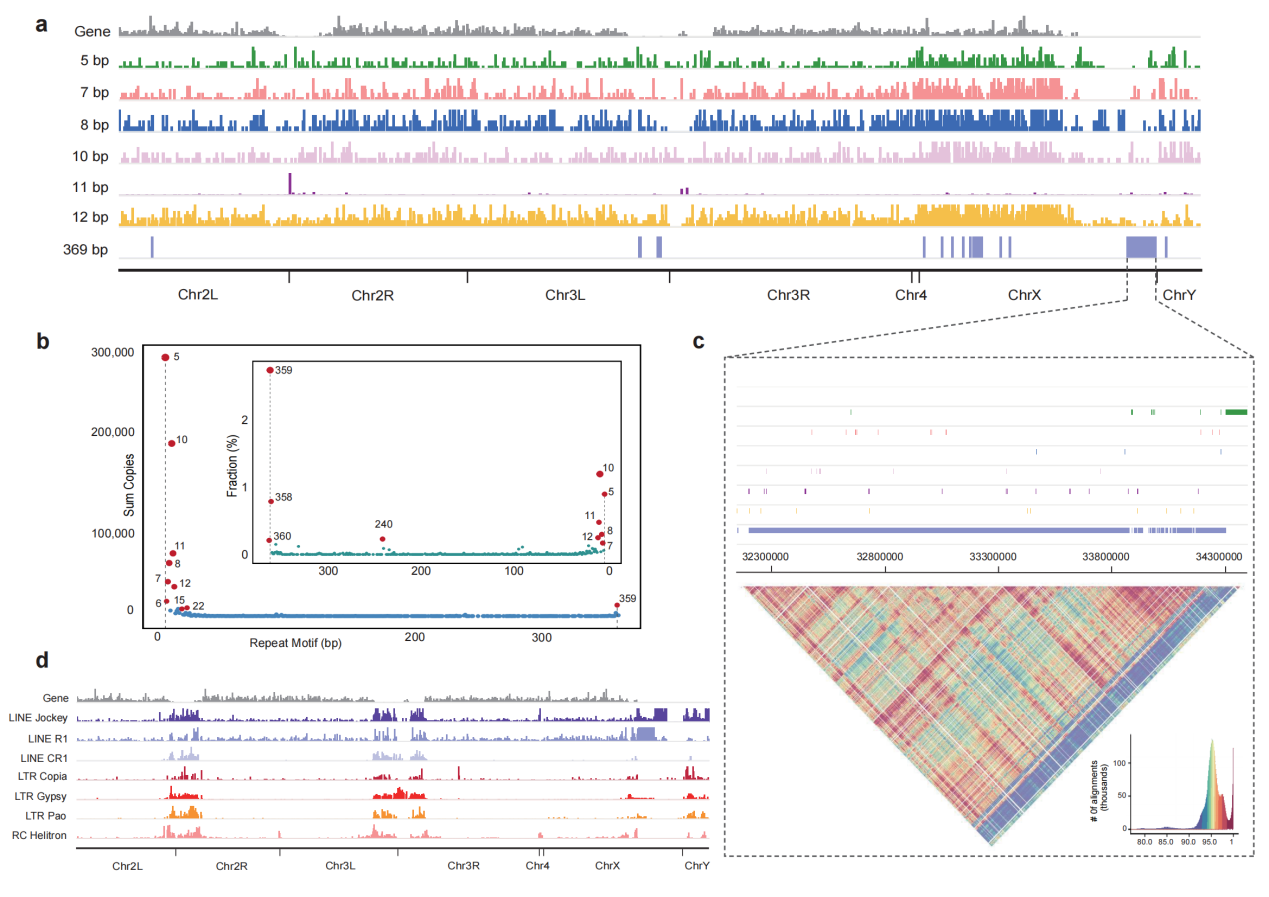
图3.基于串联重复与CENP-A结合的着丝粒区域鉴定结果
(4)发现并验证新基因功能:基于多证据整合的注释流程,团队在Dm.nT2T中新发现了92个基因。通过CRISPR/Cas9基因敲除技术,研究人员对其中两个组织特异性表达基因进行了功能验证:a) Chr3L.2449:在触角、唇瓣等嗅觉组织高表达。敲除该基因后,突变果蝇在Y型管嗅觉行为实验中,对厌恶气味(3-辛醇)的敏感性显著下降(图4h),首次证实该基因对果蝇嗅觉功能至关重要。该基因在R6参考基因组中位于未组装的缺口区域。b) Chr2R.722:在幼虫期高表达,敲除后虽未引起明显形态学缺陷,但转录组分析提示其可能参与几丁质鞣化、免疫等通路。
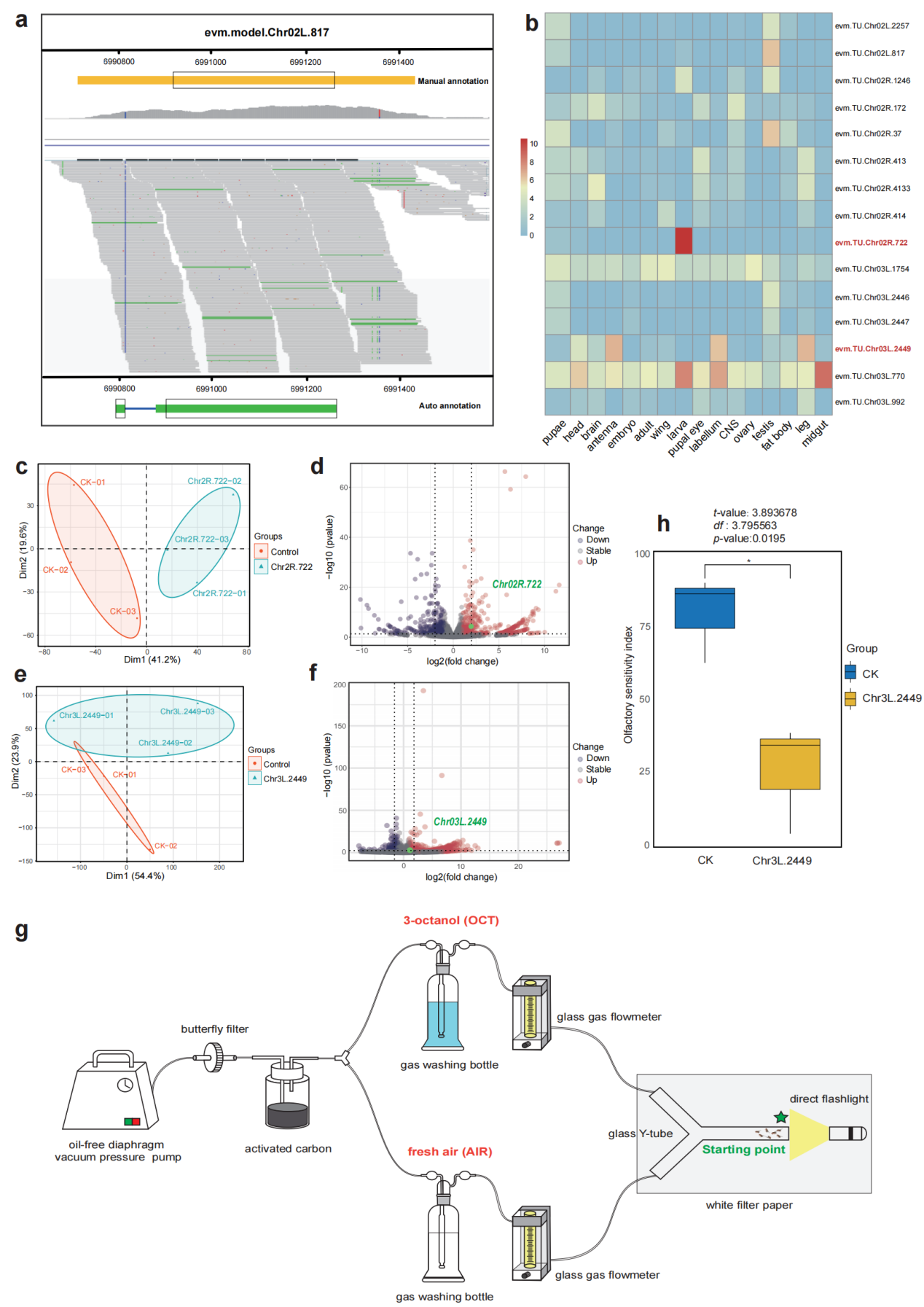
图4.新基因Chr3L.2449的组织表达谱及其敲除导致的嗅觉行为缺陷
这项研究不仅为果蝇研究领域提供了一个近乎完整的、高质量的基因组参考,更重要的是,它揭示了不同果蝇品系间存在的广泛结构变异,并证明了这些‘暗物质’区域中蕴含着具有重要生物学功能的基因,为研究种内遗传多样性、复杂性状的遗传基础以及基因组不稳定性的机制提供了不可替代的资源。未来,整合单细胞测序、表观基因组学等多组学数据,并跨多个果蝇品系进行比较基因组学研究,将进一步阐明这些新发现的结构元件与基因在进化、适应与疾病模型中的功能。
来源:生态与环境学院
编辑:张懿淼 责任编辑:李哲